Стандартные форматы представления биологических последовательностей
Задача электронного представления генетической информации встала перед исследователями, когда появилось специализированное программное обеспечение, способное обрабатывать эту информацию. Поскольку различных нуклеотидов и стандартных аминокислот немного, логично, что их стали кодировать одним символом. Обычно для кодирования нуклеотида используется первая буква в его названии. В то же время, названия многих аминокислот начинаются с одинаковых букв, поэтому коды многих аминокислот не совпадают с их первыми буквами, т.к. используются те символы, которые не заняты.
Формат FASTA
FASTA — один из наиболее популярных тестовых форматов для представления нуклеотидных и аминокислотных последовательностей, в котором нуклеотиды или аминокислоты кодируются при помощи одного символа. Данный формат был впервые использован в программном обеспечении FASTA [53], но позже стал общепринятым стандартом в области биоинформатики.
Файл в формате FASTA [16] представляет собой простой текстовый файл, первая строка которого должна начинаться с символа ״>״ или ״;״. Эта строка содержит идентификатор (имя) последовательности и некоторую вспомогательную информацию, необходимую для краткого описания последовательности. Другие строки, начинающиеся с символа ״;״ являются комментариями и игнорируются программным обеспечением. Впоследствии от использования символа ״;״ в начале файла и от дополнительных комментариев, начинающихся с этого же символа, отказались и стали использовать только символ ״>״ и одну единственную первую строку для идентификации последовательности и комментариев. При этом между знаком ״>״ и первым символом в имени последовательности не должно быть никаких вспомогательных символов или знаков.
Таблица 1.1.
Обозначение нуклеотидов в формате FASTA
| Код нуклеиновой кислоты | Значение | Обозначение |
| A | A | аденин |
| C | C | цитозин |
| G | G | гуанин |
| T | T | тимин |
| U | U | урацил |
| R | A или G | пуриновые основания |
| Y | С или T или U | пиримидиновые основания |
| K | G или T или U | - |
| M | A или C | - |
| S | C или G | Сильное взаимодействие |
| W | A или T или U | Слабое взаимодействие |
| B | не A | любое, кроме A |
| D | не C | любое, кроме C |
| H | не G | любое, кроме G |
| V | не T и не U | Любое, кроме Т и U |
| N | A или С или G или T или U | любой |
| X | замаскированный | - |
| - | неизвестная последовательность произвольной длины | - |
После строки с заголовком и комментариями начинается описание последовательности. Оно представлено в виде одной или нескольких строк с буквенным кодом. Рекомендуется, чтобы каждая строка состояла не более чем из 80 символов, хотя возможно использование и более длинных строк (до 132 символов). При кодировании последовательности нуклеотидов, буквы A, C, G, T и U кодируют, соответственно, аденин, цитозин, гуанин, тимин и урацил. Также некоторые буквы (табл. 1.1.) кодируют позиции, в которых находится один нуклеотид из некоторого множества (это используется, если точно неизвестно, какой именно нуклеотид там находится). Символы, кодирующие аминокислоты, представлены в таблице 1.2. Символ ״-״ (дефис) кодирует неизвестную последовательность произвольной длины.
Таблица 1.2.
Обозначение аминокислот и специальных триплетов
| Код | Описание |
| A | Аланин |
| B | Аспарагиновая кислота или Аспарагин |
| C | Цистеин |
| D | Аспарагиновая кислота |
| E | Глутаминовая кислота |
| F | Фенилаланин |
| G | Глицин |
| H | Гистидин |
| I | Изолейцин |
| K | Лизин |
| L | Лейцин |
| M | Метионин |
| N | Аспарагин |
| O | Пирролизин |
| P | Пролин |
| Q | Глутамин |
| R | Аргинин |
| S | Серин |
| T | Треонин |
| U | Селеноцистеин |
| V | Валин |
| W | Триптофан |
| Y | Тирозин |
| Z | Глутаминовая кислота или Глутамин |
| X | любая |
| * | остановка трансляции |
| - | неизвестная последовательность произвольной длины |
В строках последовательности допустимо использование пробелов, табуляции и символа ״*״, которые, как правило, игнорируются программным обеспечением. Пробелы и табуляция служат для выравнивания последовательностей, а символ ״*״ является признаком конца последовательности. Позже этот символ был заменен на пустую строку. Если в файле встречается несколько символов ״>״, то каждый из них будет указывать на начало новой последовательности.
Формат FASTQ
FASTQ - формат представления биологической последовательности (обычно, последовательности нуклеотидов) совместно с данными об их качестве [33].
Этот формат используется для представления данных секвенирования, так как позволяет представить как саму последовательность, так и вероятность, что каждый из элементов последовательности указан правильно. Для этого, кроме символов, кодирующих элементы последовательности, используется символы, кодирующие уровень качества.
Уровень качества — целое число в некотором диапазоне. Известно два различных способа выражать уровень качества через вероятность ошибки. Чаще всего используется следующая формула (1.1):
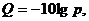 (1.1)
(1.1)
Здесь Q - уровень качества, а p - вероятность, что этот элемент последовательности ошибочный.
Реже применяется второй способ (1.2):
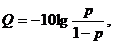 (1.2)
(1.2)
При больших значениях Q (и, соответственно, при малых p) эти способы дают практически идентичные результаты, но при малых значениях Q (Q < 13) и больших значениях p (p > 0,05) их значения заметно различаются.
Сам же файл содержит четыре строки для каждой последовательности. Первая строка начинается с символа ״@״, после которого идёт идентификатор (имя) и описание последовательности, как и в формате FASTA. Следующая строка содержит последовательность символов, кодирующих саму последовательность, также аналогично формату FASTA. Третья строка, начинающаяся с символа ״+״, после которого может идти описание последовательности (третья строка будет отличаться от первой только тем, что первый символ заменён на ״+״). Зачастую эта строка содержит лишь символ ״+״ без какого-либо описания.
Последняя строка кодирует уровни качества. Её длина равна длине второй строки, а каждый символ кодирует информацию о качестве (вероятности) элемента последовательности, закодированного соответствующим символом во второй строке. Сами же уровни кодируются таким образом: ASCII-код символа равен уровню качества плюс некоторая константа. Константа обычно имеет значение 33 или 64. В любом случае, код символа не должен превышать 127.
Формат GenBank
Формат GenBank позволяет представлять больше дополнительной информации о последовательностях [17]. Файл в формате GenBank состоит из нескольких записей, каждая из которых может занимать несколько строк. Все строки в записи, кроме первой, начинаются с пробела, это позволяет легко находить границы записей.
Каждая запись начинается с заголовка (идентификатора), за которым идёт значение (через один или несколько пробелов). Некоторые записи могут содержать подзаписи. Они форматируются аналогично записям. После этого в начало каждой строки записывается несколько пробелов. Таким образом, значения подзаписей выровнены правее значений записей, а те выровнены правее имён записей.
Имена записей имеют предопределённое значение. Например, запись с заголовком DESCRIPTION хранит описание последовательности, запись с заголовком SOURCE идентифицирует объект, из которого взята последовательность, а запись с заголовком REFERENCE (их может быть несколько) используется для описания ссылок на публикации.
Запись с заголовком ORIGIN содержит саму последовательность. Каждая строка, кроме последней, содержит 60 элементов, разбитых пробелами на группы по 10. Для представления элементов используются строчные буквы. В начало каждой строки добавляется текущая позиция, начиная с единицы, и пробел.
Помимо трех основных форматов FASTA, FASTQ и GenBank, в биоинформатике существует еще множество менее распространенных текстовых форматов представления данных биологических последовательностей, таких как: EMBL, GCG, GCG-RSF, IG и др. [15].
